自然界中存在着数量庞大的微生物,每种微生物具有特定的代谢特性。因此具有不同代谢特性的微生物成员之间可以组成功能各异的“队伍”。生物体的代谢特性是由基因组编码的蛋白(或酶)所催化,即存在着基因组-酶-代谢特性的递进关系。
近日,南京农业大学生命科学学院蒋建东教授课题组发表了新的研究成果,该研究基于基因组-酶-代谢特性的关系构建了多菌株构代谢模型,揭示了基于多菌株代谢模型可以设计微生物“队伍”,从而获得最佳的污染物降解菌群。也就是说,多菌株代谢模型可以作为微生物菌群的“教练”,在充分了解“队员”的代谢特征后,设计出高效降解污染物的“队伍”。
自然环境中的微生物大多是以菌群的形式存在,菌群成员之间也存在复杂的相互作用关系,这些菌群在影响生态可持续发展和人类健康的地球生物化学循环(包括元素循环、污染物降解、食品发酵等)过程中发挥着重要作用。比如,有机污染物的微生物降解大多是由微生物菌群中各成员催化的互补或重叠的降解过程来行使,而很少是由单个菌株独立完成。因此,微生物菌群的组成和菌群成员之间的相互作用关系不仅影响污染物的代谢速率,而且决定了污染物的最终产物。然而,人们对污染物的降解效率、微生物菌群结构、环境条件(如营养条件等)之间的关系认识还很缺乏。因此目前微生物菌群应用的实施途径还主要依靠经验和不断的试错实验,缺乏从头设计微生物“队伍”的工具。
为了弄清微生物菌群、功能和环境的关系,寻找可以设计具有特定功能的微生物“队伍”的“教练”,南京农业大学生命科学学院蒋建东教授团队与以色列农业研究组织(ARO) Shiri Freilich博士合作,利用高通量测序技术鉴定阿特拉津降解菌群的“队员”构成,基于基因组编码的代谢网络明确各“队员”的代谢特性,通过多菌株代谢模型模拟不同“队员”组成的“队伍”的阿特拉津降解功能,设计出高效降解阿特拉津的“队伍”,阐明具有直接降解功能的菌株与其他菌株之间的合作(代谢物质交换)提高了阿特拉津的降解效率,揭示了除草剂阿特拉津降解菌群的结构、功能与相互作用关系,相关结果以“Modeling microbial communities from atrazine contaminated soils promotes the development of biostimulation solutions”为题,在线发表在国际微生物生态学会会刊The ISME Journal (IF5yr=11.663)上(https://www.nature.com/articles/s41396-018-0288-5)。该论文第一署名单位为南京农业大学,生科院徐希辉副教授和以色列农业研究组织的Raphy Zarecki博士为共同第一作者,蒋建东教授和ShiriFreilich博士为共同通讯作者。
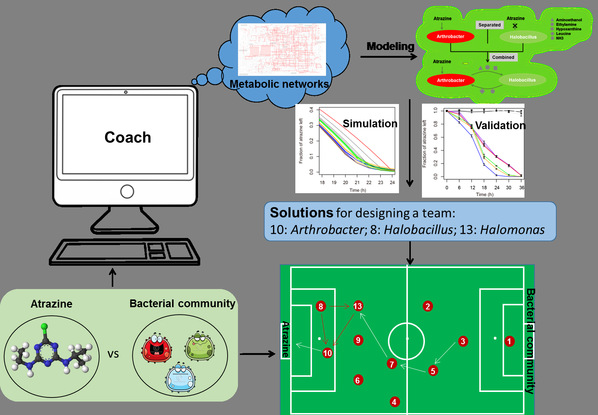
多菌株代谢模型设计微生物菌群
该研究采用多菌株代谢模型,模拟并比较了包含阿特拉津降解菌株Arthrobacter和其余4个无降解功能菌株的16种不同组合方式的菌群,表明多菌株的菌群组合在阿特拉津降解及生物量方面的表现明显好于单菌株Arthrobacter,最佳菌群即Arthrobacter与Halobacillus和/或Halomonas的组合,菌群降解效果优的机制在于Halobacillus利用Arthrobacter降解阿特拉津产生的物质(如氨基乙醇,乙胺和次黄嘌呤)生长,并反馈代谢产物(如氨和亮氨酸)供Arthrobacter利用,促进了Arthrobacter的生长和对阿特拉津的降解。此外,外加碳源可以明显促进菌群的阿特拉津降解速度。这些模型预测结果得到了实验室生长降解实验和土壤盆鉢降解实验的有效验证。值得注意的是,虽然在阿特拉津处理土壤中Halobacillus的丰度显著提高,但是在该土壤的富集液中并没有检测到Halobacillus,说明基因组代谢模型可以弥补传统富集分析方法的局限。该研究不仅揭示了微生物菌群高效代谢阿特拉津的机制,而且表明测序技术结合代谢模型构建可以基于计算机模拟微生物菌群结构和功能,在最佳功能菌群的人工设计方面具有很大的应用前景。




